|
つくば生物ジャーナル Tsukuba Journal of Biology (2005) 4:
TJB200501200100762
ポリエステル系生分解性プラスチック分解酵素遺伝子の解析
佐藤 愛美 (筑波大学 生物学類 4年) 指導教員: 中島 敏明 (筑波大学 生命環境科学研究科)
[目的]
近年、自然循環型の素材であり、非蓄積性で環境負荷の少ない素材特性を持っている生分解性プラスチックの利用に高い関心が寄せられており、市場も順調に拡大しつつある。廃生分解性プラスチック混合物のモノマー、オリゴマーへの段階的分解とその後の回収、再利用の実現に向け、各生分解性プラスチックに基質特異性を有する酵素及び酵素遺伝子の解析が必要となる。当研究室において分離された3A株は、各種ポリエステル系生分解性プラスチックに対して高い分解能を示す。本研究では3A株由来の分解酵素を生分解性プラスチックのバイオケミカルリサイクルプロセスに応用することを目的として、3A株の菌学的諸性質の解明及び分解遺伝子の取得を試みた。
[方法及び結果]
1. 16S rDNA塩基配列を指標とした3A株の同定
3A株のシングルコロニーを単離し、ダイレクトコロニーPCR法により16S rDNAの全長を増幅した。PCR産物をpGEM-Tベクターにligationし、形質転換体のプラスミドを抽出した。インサートDNAの塩基配列を決定し、得られた塩基配列をBLASTに供し相同性検索を行い、系統樹を作成した。3A株の16S rDNA 5’側640bpをBLASTに供したところ、Leptothrix mobilisの相同性が95%であり、3A株はLeptothrix属の新種である可能性が高いことが分かった。
2.ショットガンクローニング法による分解酵素遺伝子のクローニング
供試生分解性プラスチックとして、ポリブチレンサクシネート-co-アジペート(PBSA)を用いた。3A株の全DNAを抽出し、Sau3AIにて限定分解を行った。約2〜5kbのDNA断片を回収し、大腸菌ベクターpUC18に組み込み、PBSAエマルジョンを加えた培地上で、コロニーの周囲にクリアゾーンを作る形質転換体を取得した。この形質転換体の有するプラスミドをpBSL1とした。
3.pBSL1の塩基配列決定
サブクローニングを行った後、最少領域の塩基配列を決定したところ、849bpのORFが確認された。この遺伝子がコードする酵素は分子量約3万で、N末端側にアミノ酸24個からなるシグナルペプチドを有していた。切断点より後方10個のアミノ酸配列は、3A株由来精製PBSA分解酵素のN末端アミノ酸配列と完全に一致した。大腸菌にて大量発現させた組み換え酵素は、PBSAフィルムを完全に分解した。相同性検索の結果、本遺伝子は既知の遺伝子とは相同性を示さない新規遺伝子であった。
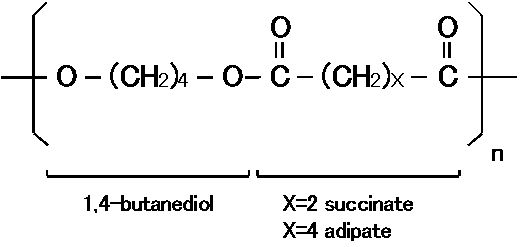
fig1. ポリブチレンサクシネート-coアジペートの構造
©2005 筑波大学生物学類
| 
