|
つくば生物ジャーナル Tsukuba Journal of Biology (2005) 4:
TJB200501200100766
転写因子MafGのDNA認識機構の解明−MafG(21-123)/DNA複合体のX線結晶構造解析
高川 紘明 (筑波大学 生物学類 4年) 指導教員:
田中 俊之 (筑波大学 生命環境科学研究科)
<序論>
Maf群タンパク質は、DNA認識に関わる塩基性領域と、二量化に必要なロイシンジッパーからなる塩基性ロイシンジッパー(bZip)構造を持つ転写調節因子であるが、bZipの直前に、EHR(extended homology region)と呼ばれる固有のアミノ酸配列を持つ(図1)。Maf群タンパク質は、このEHRと塩基性領域によって、MAREと呼ばれる13-14 bpのDNA配列を特異的に認識する。MAREは、中央にAP1-core(FosやJunなどのbZipタンパク質が認識する配列)を持ち、その両端に3 bpからなる flanking 領域が存在する(図1)。Maf群タンパク質は、他のbZipタンパク質と同様に塩基性領域でAP1-coreと結合するが、同時に固有のEHRでflanking領域を認識し、MAREと強く結合すると考えられている。Maf群タンパク質の一つであるMafGのDNA結合ドメイン(EHRと塩基性領域からなる)の構造は、既に当研究室で決定したが、具体的にどのような相互作用でMAREを認識しているかは未だ明らかになっていない。そこで、Maf群タンパク質のDNA認識機構を解明するために、EHRとbZipを持つMafG(21-123)と、MAREを含むDNAとの複合体のX線結晶構造解析を行った。
<方法>
(1) MafG(21-123)の調製
当研究室で既に確立されている大腸菌を用いた発現と精製のプロトコールに従って、MafG(21-123)の大量発現と精製を行った。
(2) MAREを含む二本鎖DNAの調製
陰イオン交換カラムと脱塩カラムを用いて精製した合成オリゴヌクレオチド 15a(5'-GTGCTGACTCATCAG-3')と15b(5'-CTGATGAGTCAGCAC-3')をアニーリングし、二本鎖DNAを得た。
(3) MafG(21-123)/DNA複合体の結晶化
MafG(21-123)と二本鎖DNAをモル比で2 : 1となるように混合して調製した複合体溶液(0.2-0.4 mM)を用い、蒸気拡散ハンギングドロップ法で結晶の作製を試みた。
(4) X線回折データの収集と解析
作製した結晶のX線回折データは、高エネルギー加速器研究機構(PF-AR, NW12)において収集した。X線の波長は0.978 Å、検出器はADSC Quantum 210, データ処理にはプログラムHKL2000を用いた。
<結果と考察>
LB培地1Lから約25 mgの精製MafG(21-123)が得られた。このタンパク質と二本鎖DNAからなる複合体について、市販のスクリーニングキットを用いて、20℃で結晶化を試みた。その結果、2-Methyl-2,4-pentanediolとPolyethylene glycol monomethylether 550を沈殿剤とする条件で微小結晶が得られたので、この条件を基に結晶化条件を最適化し、より大きいサイズ(約0.5 mm × 0.05 mm × 0.1 mm)の単結晶を得ることに成功した(図2)。得られた六方晶の結晶を用いて、分解能約4 ÅのX線回折データを収集したところ、格子定数はa = b = 180 Å、c = 409 Åであった。現在、このデータを用いて、MafGのDNA結合ドメインのNMR構造をサーチモデルとした分子置換法による位相決定を試みている。また、より高分解能のデータを得るために、結晶化条件や結晶化に使用するDNAの長さなどについて検討中である。
<謝辞>
本研究を行うにあたり、多くの御指導と御協力を頂いた東北大学 多元物質科学研究所 黒河博文博士に深く感謝致します。
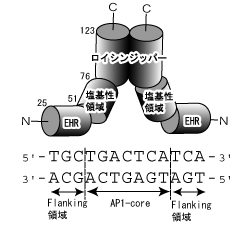
図1 Maf群タンパク質のDNA結合領域とその認識配列
NとCはそれぞれN末端とC末端を、数字はMafG
におけるアミノ酸残基の番号を示す。

図2 MafG(21-123)/DNA複合体の結晶写真
©2005 筑波大学生物学類
|

