|
つくば生物ジャーナル Tsukuba Journal of Biology (2006) 5: TJB200601200200766
RNAi法によるショウジョウバエキノコ体遺伝子の機能解析
中島 大樹(筑波大学 生物学類 4年) 指導教員:古久保-徳永 克男(筑波大学 生命環境科学研究科)
導入・目的
キノコ体はショウジョウバエをはじめとする多くの節足動物に存在する発達した神経構造であり、学習・記憶・認知の中枢として機能している。キノコ体は細胞体であるケニオン細胞から軸索が束になって伸び、これらの束は前方で特徴的な5つのローブを形成する。これまでに我々の研究室において、ショウジョウバエのゲノム遺伝子をほぼ完全に網羅したマイクロアレイを用い、キノコ体で発現が亢進する多数の遺伝子が同定されてきた。本研究は、同定されたこれらの遺伝子のキノコ体における発現をRNA干渉法(RNAi法)により抑制し、それぞれの遺伝子がどのようにキノコ体形成に関っているか解明する足がかりを築くことを目的としている。
材料・方法
実験に用いたRNAi系統は、国立遺伝学研究所から分与していただいた系統を使用した。これらの系統はショウジョウバエのそれぞれに遺伝子に対するRNAi配列がUAS配列の下流に組み込まれている。この系統を、キノコ体と眼特異的なGal4エンハンサートラップOK107と、UASの下流に膜結合型GFPを結合させた配列を併せ持つ系統(UAS-mCD8::GFP ; OK107)と掛け合わせ、遺伝的にRNAiとヘテロになるようにF1を作成し、その成虫脳を共焦点顕微鏡で観察した。この交配によりGal4を発現させ、RNAiを、複眼とキノコ体で特異的に誘導するとともに、遺伝子発現阻害による全般的な致死性を減少させることができる。さらに同時に、キノコ体や複眼の構造をGFPで可視化することが出来る。今回、酵素や転写因子などの機能のわかっている遺伝子から、機能未知の遺伝子まで、約40のRNAi系統をUAS-mCD8::GFP ; OK107と掛け合わせ、その影響を観察した。それぞれの遺伝子について、同じRNAi配列を異なる染色体上にもつ形質転換系統が2つ検討し、全部で約80系統を観察した。また、1つの系統においては、約20個体の脳を解剖観察し、キノコ体の各部位における異常の度合いを−,±,+,++としてランク付けし、さらにそれぞれのランクに応じて(0,1,2,3点)と点数をつけ、その点数の合計をこの個体の異常の度合いを示す指数として用いた。
結果・考察
いくつかの系統で、キノコ体の形体異常が見られた。特にcGMP特異的ホスホジエステラーゼをコードしている遺伝子CG9296のRNAi系統においてはキノコ体の形体異常だけでなく、頭部や複眼の形体が異常になる強い形質が観察された。マイクロアレイの際に特に差の出た14遺伝子が決定されており、今回そのうちの7系統観察を行ったが、顕著な形体異常の傾向は観察されなかった。また多くの系統において20個体中1〜2個強い形体異常が観察されたが、その系統の中で一定の傾向を持った形質が観察されることは少なかった。今回の実験ではRNAiが本当に効いており、実際に特定の遺伝子発現が抑えられているかの確認はしておらず、実際に発現をある程度抑制していたとしても、形質として現れなかった可能性もあるので、まだまだ実験結果としては不完全であるが、キノコ体形成遺伝子の今後の研究を行う上での指標として、重要な基礎情報となると考えられる。
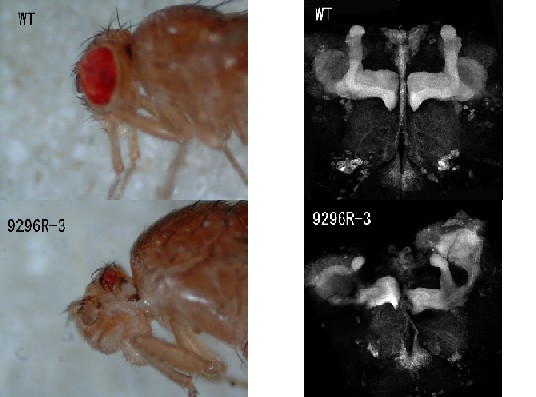
図1.ショウジョウバエ頭部の側方実体写真(左)とGFPにより可視化されたキノコ体を正面から観察した写真(右)
・上段の2つはコントロール
・下段の2つはCG9296の発現をRNAiで抑制した系統(9296R-3):WTと比較して、頭部及びキノコ体の形
体が異常である
©2006 筑波大学生物学類
|